Tracking software that works
Many of the projects in this SFB initiative involve, or even rely on, cell or particle tracking. Quantitative results are of paramount importance to establish and understand differences arising from various stimuli or mutations. Unfortunately, existing tracking methods either lack an incarnation in terms of usable software, or are overfitted to a particular use case and do not cope well with variations in the experimental setup or with biological variability. Reliable automated tracking is still unavailable in most circumstances.
Achieved goals of this project:
(i) implemented tracking with learning software that can be trained by the end user allowing adjusting to changing experimental conditions;
(ii) developed an interface to Fiji / Mamut for proofreading of automated predictions
(iii) deployed this software within the SFB and evaluated its robustness and usability;
(iv) trained the SFB experimental groups to operate this software independently; and
(v) deployed the program worldwide as an open source software.
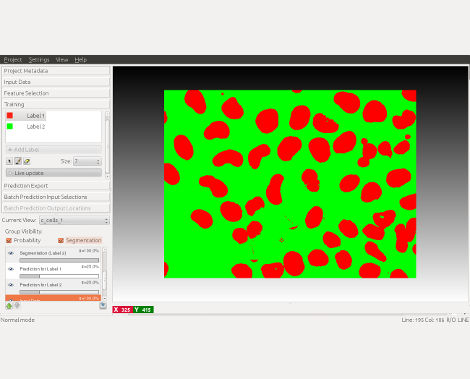
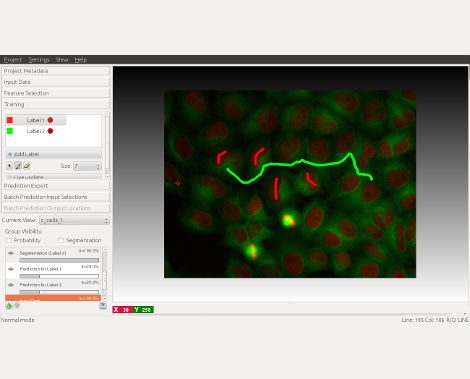
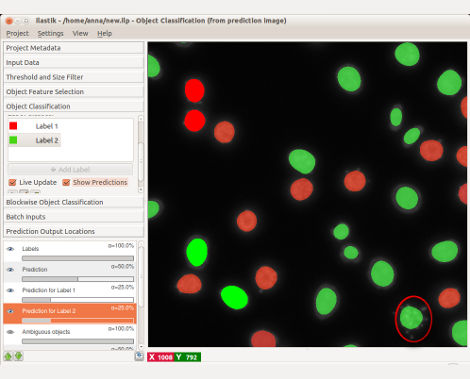
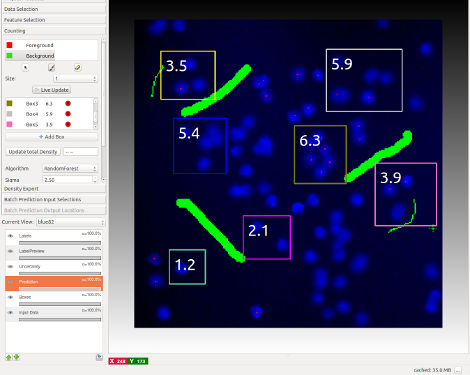
On the technical side, we have packaged structured learning in a program that is accessible to the end user, aiming for an interactive workflow that takes the user in the loop during the training phase. On the software side, we have built on open source packages for image processing and machine learning, drawing especially on the “just in time” computation paradigm of ilastik to afford interactive response times even on very large data sets. The open source tracking with learning program has facilitated the analysis of new biological experiments and is making quantitative large-scale analyses possible in this SFB and beyond.
For more information: http://www.ilastik.org
Viele Projekte in diesem SFB sind darauf angewiesen, Zellen oder andere Partikel über die Zeit zu verfolgen (“Tracking”). Die quantitative Analyse der resultierenden Trajektorien ist von höchstem Interesse, um zu verstehen, wie unterschiedliche experimentelle Bedingungen die Bewegungen von Zellen oder Partikeln beeinflussen. Leider sind existierende Trackingmethoden entweder nicht in einfach benutzbaren Softwarepaketen verfügbar, oder sie sind so sehr auf einen bestimmten Anwendungsfall spezialisiert, dass sie sich nicht adäquat an wechselnde Bedingungen und die biologische Vielfalt anpassen lassen. Eine verlässliche Automatisierung des Trackings ist in den meisten Anwendungen bisher unmöglich.
Das vorliegende Projekt verfolgt deshalb vier Ziele:
(i) Implementation einer Tracking-Software, die jeder Endbenutzer mittels maschinellem Lernen an neue Experimente anpassen kann;
(ii) Anwendung dieser Software innerhalb des SFB und Optimierung ihrer Verlässlichkeit und Benutzbarkeit;
(iii) Schulung der Experimentatoren im SFB, damit sie diese Software selbständig einsetzen können; und
(iv) weltweite Freigabe der Software als Open-Source-Projekt.
Den technischen Kern der Software wird das sogenannte “strukturierte Lernen” bilden, das in einer interaktiven Umgebung so präsentiert werden soll, dass der Benutzer während der Trainingsphase des Algorithmus laufend Feedback über die aktuelle Analysequalität erhält und jederzeit korrigierend eingreifen kann (“user in the loop”). Die Software wird auf existierenden Open-Source-Paketen für Bildverarbeitung und maschinelles Lernen aufbauen, insbesondere auf ilastik, dessen “just in time”-Paradigma auch auf sehr großen Datensätzen interaktive Antwortzeiten ermöglichen wird. Die frei zugängliche Trackingsoftware wird den Experimentatoren die derzeitige aufwändige manuelle Analyse abnehmen und soll dadurch die quantitative Analyse sehr großer Datensätze im Routinebetrieb innerhalb dieses SFBs und darüber hinaus ermöglichen.
Weitere Informationen: http://www.ilastik.org
National Collaborations
Denk ,Winfried, Max Planck Institute, Heidelberg
Draguhn, Andreas, University of Heidelberg
Hufnagel, Lars, EMBL, Heidelberg
Wittbrodt, Jochen, University of Heidelberg
International Collaborations
Bock, Davi, HHMI Janelia Farm, Ashburn, USA
Branson, Kristin, HHMI Janelia Farm, Ashburn, USA
Cardona, Albert, HHMI Janelia Farm, Ashburn, USA
Cook, Matthew, ETH Zurich, Switzerland
Gerlich, Daniel, Institute of Molecular Biotechnology, Vienna, Austria
Knott, Graham, Lausanne, Switzerland
Nadler, Boaz, Weizmann Institute, Rehovot, Israel
Scheffer, Lou, HHMI Janelia Farm, Ashburn, USA
Steen, Hanno, Boston Children’s Hospital, Boston, USA
Steen, Judith, Boston Children’s Hospital, Boston, USA
Zlatic, Marta, HHMI Janelia Farm, Ashburn, USA
.
.
.

.
Project Staff

Janez Ales, PostDoc

